Ensayos clínicos aleatorizados con R
En el trabajo habitual, debemos decidir en numerosas ocasiones y para multitud de intervenciones qué individuos o qué corrales elegimos. Por ejemplo, debemos tomar 30 muestras de sangre de un cebadero de cerdos que tiene 100 corrales. En el mejor de los casos, elegimos un cerdo cualquiera de un corral y continuamos eligiendo cualquier cerdo cada 3 corrales. Al final habremos realizado un muestreo dirigido sistemáticamente. ¿Es correcta esta forma de elegir a los corrales y a los cerdos?
Bueno, es mejorable.
La mejor opción sería elegir los corrales mediante números aleatorios, es decir, llevar a cabo los conocidos «ensayos aleatorizados».
Pero, ¿qué son los números aleatorios?
Podríamos definirlos como aquellos que dependen del azar o casualidad y que tienen la misma probabilidad de aparecer que cualquier otro del conjunto. Todos tenemos implícitamente la idea de azar, dado que la lotería es un juego de azar en la que se eligen números aleatoriamente.
¿Cómo podemos obtener números aleatorios con R?
En este post, vamos a realizar los cálculos del ejemplo propuesto utilizando lenguaje de programación R.
Es sumamente sencillo obtener muestras aleatorias de un conjunto de casos.
Imaginemos que queremos obtener tres números aleatorios. Por definición, los números aleatorios siempre están en el intervalo entre 0 y 1. Si abrimos R y, sin cargar la librería Rcmdr que nos da acceso a nuestro RCommander, escribimos directamente en la consola:
runif(3) [1] 0.4326041 0.1943911 0.2656029
Ya los tenemos. Luego con ellos podemos hacer o calcular lo que hubiéramos pensado (después continuaremos con el ejemplo).
Si el lector hace esta misma operación no obtendrá los mismos números aleatorios, debido precisamente al azar.
Sin embargo, en ocasiones necesitamos obtener los mismos números aleatorios. Para ello, debemos “sembrar” una semilla de aleatorización que le indica al generador de números aleatorios desde dónde debe comenzar a obtenerlos; así cada vez que queramos ejecutar la rutina obtendremos el mismo resultado.
Veámoslo con un ejemplo.
Si escribimos:
set.seed(1) runif(3)
obtendremos:
[1] 0.2655087 0.3721239 0.5728534
Si ahora queremos obtener los mismos números aleatorios, de nuevo escribimos:
set.seed(1) runif(3)
y vemos que los números obtenidos son siempre los mismos.
¿Cómo nos ayudan los números aleatorios en los ensayos aleatorizados?
Sigamos con la idea inicial. Queremos obtener 30 muestras de sangre de una granja que tiene 100 corrales. ¿De qué corrales debo obtener las muestras?
Lo primero que haremos es decirle a R que tenemos 100 corrales y los numeraremos del 1 al 100 colocándolos en un vector. Primero lo definimos y después le pedimos que lo escriba:
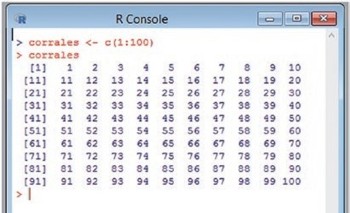
Ahora, simplemente le decimos que queremos una lista de 30 corrales aleatoriamente, con lo que obtendremos:
> sample(corrales, 30) [1] 91 20 89 92 64 60 6 99 17 63 35 69 44 100 86 33 66 78 18 53 11 22 31 2 30 84 26 36 88 73
Si queremos una segunda lista podemos escribirlo de nuevo y obtenemos:
> sample(corrales, 30) [1] 19 82 66 78 11 69 39 77 60 72 50 48 70 3 42 63 59 40 71 36 20 6 8 25 83 90 31 67 22 33
Pero si queremos tener reproducibilidad, es decir, que nos salga lo mismo para enseñarlo o hacer algo con el resultado, tendremos de nuevo que sembrar una semilla de aleatorización. Así:
set.seed(123) sample(corrales, 30) [1] 29 79 41 86 91 5 50 83 51 42 87 98 60 94 9 77 21 4 27 78 72 55 90 85 81 54 89 44 84 11 set.seed(123) sample(corrales, 30) [1] 29 79 41 86 91 5 50 83 51 42 87 98 60 94 9 77 21 4 27 78 72 55 90 85 81 54 89 44 84 11
Esta forma de obtener casos aleatorios se denomina sin reemplazo, ya que cada vez que un caso es elegido ya no puede, en esa ocasión, ser elegido de nuevo. Ahora ya sabemos en qué corrales debemos muestrear. Si tuviéramos identificados uno a uno los cerdos, podríamos hacer lo mismo dentro de cada corral.
Elección aleatoria de granjas para nuestros ensayos aleatorizados
¿Podríamos hacer lo mismo con el nombre de granjas si queremos, por ejemplo, elegir un grupo de ellas al azar para la realización de una prueba? Por supuesto que sí. Definamos primero el vector que contendrá las granjas. Por ejemplo:
granjas <- c(“Rubiales”, “Coronas”, “García”, “Mallén”, “Coromina”, “Mateo”, “Sahún”, “Benedicto”, “Daniela”, “Pérez”)
Como R es un entorno a veces poco amigable, se recomienda escribir las comillas directamente en la consola, ya que si no, puede retornar un error del tipo “Error: unexpected input in “granjas <- c(“““)”. Si ahora escribimos en la consola “granjas”, aparecerá nuestro listado. Imaginemos que necesitamos elegir a cinco de ellas. Sembraremos una semilla de aleatorización (aunque no es necesaria, sí es conveniente para repetir los resultados si los necesitáramos por cualquier motivo):
set.seed(43) sample(granjas, 5) [1] “Coromina” “Daniela” “Rubiales” “Pérez” “Coronas”
Ya tenemos el conjunto de granjas listo.
Un paso más, la librería «dplyr»
R tiene muchas librerías que habilitan la función de muestreo (sampling). Una de las más famosas por su utilidad en el manejo de datos es la libre- ría “dplyr”. Si no la tenemos instalada, lo primero será obtenerla yendo a “Paquetes/Instalar paquetes”, donde elegimos, por ejemplo, el repositorio 0-Cloud (https) y luego la buscamos en la lista que aparece. Una vez instalada, la tenemos que cargar mediante “Paquetes/Cargar paquete…” y elegimos “dplyr”.
Cargamos primero la librería dplyr con:
library(dplyr)
Posteriormente debemos transformar el vector “corrales” en un data frame mediante:
corrales <- as.data.frame(corrales)
Sembramos de nuevo la semilla:
set.seed(123)
Y pedimos los 30 corrales con:
sample_n(corrales, 30)
y obtendremos la misma lista que antes.
¿Cuál es la ventaja de usar la librería dplyr?
Si los datos provienen, por ejemplo, de una base de datos, los cargaremos en R y serán un objeto denominado data.frame. El muestreo se puede realizar en el mismo objeto. Además, la lista obtenida se puede guardar en un fichero, asignando el resultado a un objeto. En nuestro ejemplo, y como queremos tomar muestras de sangre, podemos denominar al archivo “Listado sangre”, luego lo ejecutamos y lo guardamos (cuidado de nuevo con las comillas):
Listado sangre <- sample_n(corrales, 30)
write.csv2 (Listado sangre, file = “Listado sangre.csv”)
R guardará el archivo creado (Listado sangre.csv) en el directorio que por defecto tengamos en nuestras preferencias. Yendo a “Archivo/Cargar dir…” podemos ver el directorio actual de trabajo y cambiarlo antes de guardar nuestro nuevo fichero.
Esperamos que os haya gustado el artículo. Si tenéis cualquier duda, podéis contactar a nuestro equipo o dejarnos un comentario debajo del post.